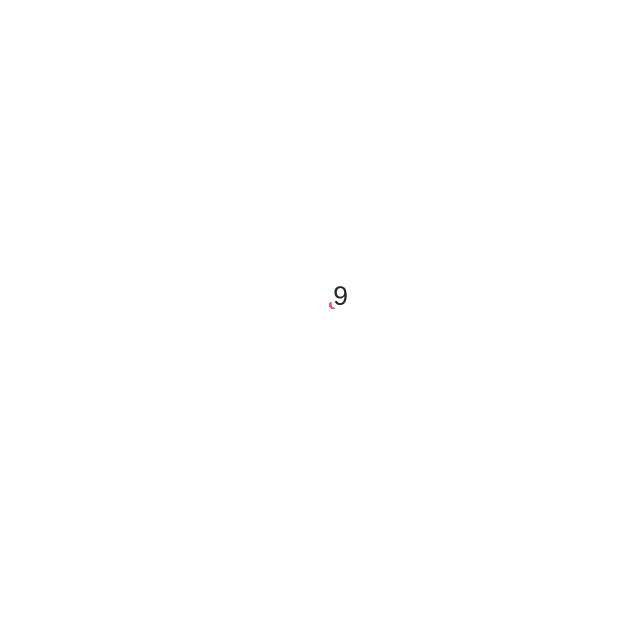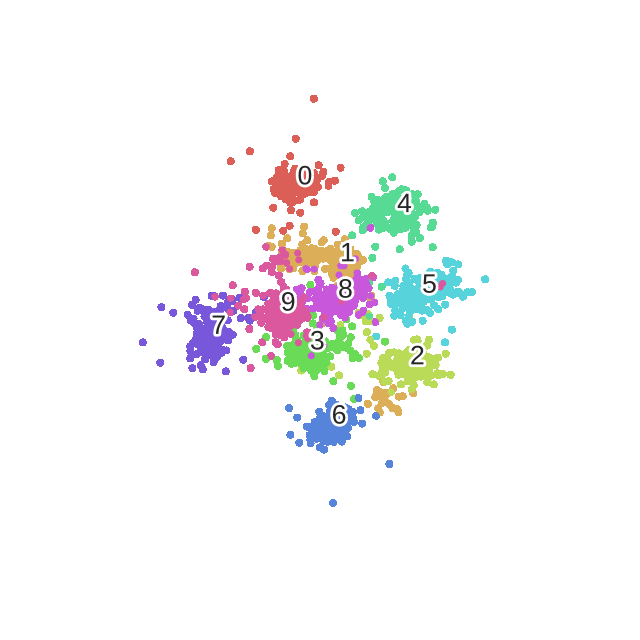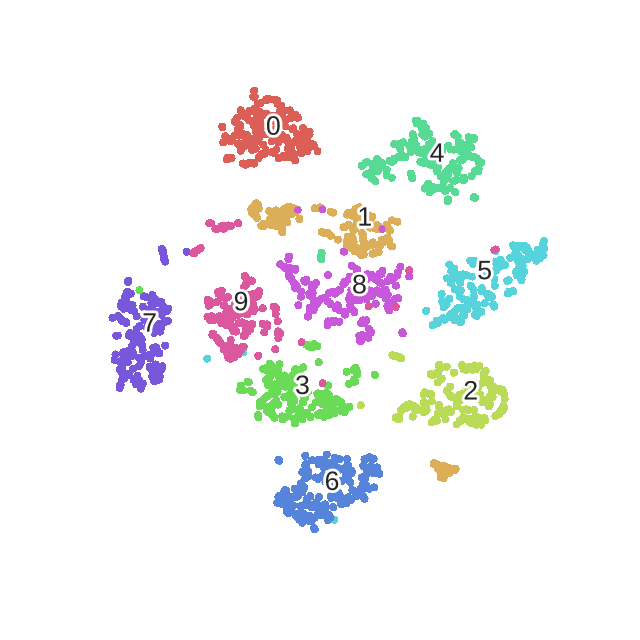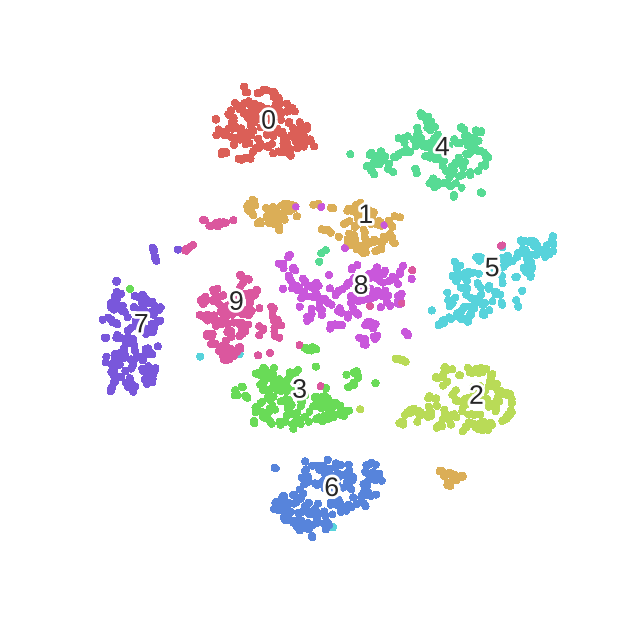t-SNE完整笔记 (附Python代码)
source link: https://blog.51cto.com/u_14682436/8923865
Go to the source link to view the article. You can view the picture content, updated content and better typesetting reading experience. If the link is broken, please click the button below to view the snapshot at that time.

t-SNE完整笔记 (附Python代码)
精选 原创t-SNE(t-distributed stochastic neighbor embedding)是用于降维的一种机器学习算法,是由 Laurens van der Maaten 和 Geoffrey Hinton在08年提出来。此外,t-SNE 是一种非线性降维算法,非常适用于高维数据降维到2维或者3维,进行可视化。
t-SNE是由SNE(Stochastic Neighbor Embedding, SNE; Hinton and Roweis, 2002)发展而来。我们先介绍SNE的基本原理,之后再扩展到t-SNE。最后再看一下t-SNE的实现以及一些优化。
1.SNE
1.1基本原理
SNE是通过仿射(affinitie)变换将数据点映射到概率分布上,主要包括两个步骤:
- SNE构建一个高维对象之间的概率分布,使得相似的对象有更高的概率被选择,而不相似的对象有较低的概率被选择。
- SNE在低维空间里在构建这些点的概率分布,使得这两个概率分布之间尽可能的相似。
我们看到t-SNE模型是非监督的降维,他跟kmeans等不同,他不能通过训练得到一些东西之后再用于其它数据(比如kmeans可以通过训练得到k个点,再用于其它数据集,而t-SNE只能单独的对数据做操作,也就是说他只有fit_transform,而没有fit操作)
1.2 SNE原理推导
SNE是先将欧几里得距离转换为条件概率来表达点与点之间的相似度。具体来说,给定一个N个高维的数据 (x_1, … , x_N)(注意N不是维度), t-SNE首先是计算概率(p_{ij}),正比于(x_i)和(x_j)之间的相似度(这种概率是我们自主构建的),即:
[{p_ {j \mid i} = \frac{\exp(- \mid \mid x_i -x_j \mid \mid ^2 / (2 \sigma^2_i ))} {\sum_{k \neq i} \exp(- \mid \mid x_i - x_k \mid \mid ^2 / (2 \sigma^2_i))}}]
这里的有一个参数是(\sigma_i),对于不同的点(x_i)取值不一样,后续会讨论如何设置。此外设置(p_{x \mid x}=0),因为我们关注的是两两之间的相似度。
那对于低维度下的(y_i),我们可以指定高斯分布为方差为(\frac{1}{\sqrt{2}}),因此它们之间的相似度如下:
[{q_ {j \mid i} = \frac{\exp(- \mid \mid x_i -x_j \mid \mid ^2)} {\sum_{k \neq i} \exp(- \mid \mid x_i - x_k \mid \mid ^2)}}]
同样,设定(q_{i \mid i} = 0).
如果降维的效果比较好,局部特征保留完整,那么 (p_{i \mid j} = q_{i \mid j}), 因此我们优化两个分布之间的距离-KL散度(Kullback-Leibler divergences),那么目标函数(cost function)如下:
[C = \sum_i KL(P_i \mid \mid Q_i) = \sum_i \sum_j p_{j \mid i} \log \frac{p_{j \mid i}}{q_{j \mid i}}]
这里的(P_i)表示了给定点(x_i)下,其他所有数据点的条件概率分布。需要注意的是KL散度具有不对称性,在低维映射中不同的距离对应的惩罚权重是不同的,具体来说:距离较远的两个点来表达距离较近的两个点会产生更大的cost,相反,用较近的两个点来表达较远的两个点产生的cost相对较小(注意:类似于回归容易受异常值影响,但效果相反)。即用较小的 (q_{j \mid i}=0.2) 来建模较大的 (p_{j \mid i}=0.8), cost=(p \log(\frac{p}{q}))=1.11,同样用较大的(q_{j \mid i}=0.8)来建模较大的(p_{j \mid i}=0.2), cost=-0.277, 因此,SNE会倾向于保留数据中的局部特征。
思考:了解了基本思路之后,你会怎么选择(\sigma),固定初始化?
下面我们开始正式的推导SNE。首先不同的点具有不同的(\sigma_i),(P_i)的熵(entropy)会随着(\sigma_i)的增加而增加。SNE使用困惑度( perplexity)的概念,用二分搜索的方式来寻找一个最佳的(\sigma)。其中困惑度指:
[Perp(P_i) = 2^{H(P_i)}]
这里的(H(P_i))是(P_i)的熵,即:
[H(P_i) = -\sum_j p_{j \mid i} \log_2 p_{j \mid i}]
困惑度可以解释为一个点附近的有效近邻点个数。SNE对困惑度的调整比较有鲁棒性,通常选择5-50之间,给定之后,使用二分搜索的方式寻找合适的(\sigma)
那么核心问题是如何求解梯度了,目标函数等价于(\sum \sum - p log(q))这个式子与softmax非常的类似,我们知道softmax的目标函数是(\sum -y \log p),对应的梯度是(y - p)(注:这里的softmax中y表示label,p表示预估值)。 同样我们可以推导SNE的目标函数中的i在j下的条件概率情况的梯度是(2(p_{i \mid j}-q_{i \mid j})(y_i-y_j)), 同样j在i下的条件概率的梯度是(2(p_{j \mid i}-q_{j \mid i})(y_i-y_j)), 最后得到完整的梯度公式如下:
[\frac{\delta C}{\delta y_i} = 2 \sum_j (p_{j \mid i} - q_{j \mid i} + p_{i \mid j} - q_{i \mid j})(y_i - y_j)]
在初始化中,可以用较小的(\sigma)下的高斯分布来进行初始化。为了加速优化过程和避免陷入局部最优解,梯度中需要使用一个相对较大的动量(momentum)。即参数更新中除了当前的梯度,还要引入之前的梯度累加的指数衰减项,如下:
[Y^{(t)} = Y^{(t-1)} + \eta \frac{\delta C}{\delta Y} + \alpha(t)(Y^{(t-1)} - Y{(t-2)})]</p><p>这里的(Y{(t)})表示迭代t次的解,(\eta)表示学习速率,(\alpha(t))表示迭代t次的动量。
此外,在初始优化的阶段,每次迭代中可以引入一些高斯噪声,之后像模拟退火一样逐渐减小该噪声,可以用来避免陷入局部最优解。因此,SNE在选择高斯噪声,以及学习速率,什么时候开始衰减,动量选择等等超参数上,需要跑多次优化才可以。
思考:SNE有哪些不足? 面对SNE的不足,你会做什么改进?
2.t-SNE
尽管SNE提供了很好的可视化方法,但是他很难优化,而且存在”crowding problem”(拥挤问题)。后续中,Hinton等人又提出了t-SNE的方法。与SNE不同,主要如下:
- 使用对称版的SNE,简化梯度公式
- 低维空间下,使用t分布替代高斯分布表达两点之间的相似度
t-SNE在低维空间下使用更重长尾分布的t分布来避免crowding问题和优化问题。在这里,首先介绍一下对称版的SNE,之后介绍crowding问题,之后再介绍t-SNE。
2.1 Symmetric SNE
优化(p_{i \mid j})和(q_{i \mid j})的KL散度的一种替换思路是,使用联合概率分布来替换条件概率分布,即P是高维空间里各个点的联合概率分布,Q是低维空间下的,目标函数为:
[C = KL(P \mid \mid Q) = \sum_i \sum_j p_{i,j} \log \frac{p_{ij}}{q_{ij}}]
这里的(p_{ii}),(q_{ii})为0,我们将这种SNE称之为symmetric SNE(对称SNE),因为他假设了对于任意i,(p_{ij} = p_{ji}, q_{ij} = q_{ji}),因此概率分布可以改写为:
[p_{ij} = \frac{\exp(- \mid \mid x_i - x_j \mid \mid ^2 / 2\sigma^2)}{\sum_{k \neq l} \exp(- \mid \mid x_k-x_l \mid \mid ^2 / 2\sigma^2)} \ \ \ \ q_{ij} = \frac{\exp(- \mid \mid y_i - y_j \mid \mid ^2)}{\sum_{k \neq l} \exp(- \mid \mid y_k-y_l \mid \mid ^2)}]
这种表达方式,使得整体简洁了很多。但是会引入异常值的问题。比如(x_i)是异常值,那么(\mid \mid x_i - x_j \mid \mid ^2)会很大,对应的所有的j, (p_{ij})都会很小(之前是仅在(x_i)下很小),导致低维映射下的(y_i)对cost影响很小。
思考: 对于异常值,你会做什么改进?(p_i)表示什么?
为了解决这个问题,我们将联合概率分布定义修正为: (p_{ij} = \frac{p_{i \mid j} + p_{j \mid i}}{2}), 这保证了(\sum_j p_{ij} \gt \frac{1}{2n}), 使得每个点对于cost都会有一定的贡献。对称SNE的最大优点是梯度计算变得简单了,如下:
(\frac{\delta C}{\delta y_i} = 4 \sum_j (p_{ij} - q_{ij})(y_i - y_j))
实验中,发现对称SNE能够产生和SNE一样好的结果,有时甚至略好一点。
2.2 Crowding问题
拥挤问题就是说各个簇聚集在一起,无法区分。比如有一种情况,高维度数据在降维到10维下,可以有很好的表达,但是降维到两维后无法得到可信映射,比如降维如10维中有11个点之间两两等距离的,在二维下就无法得到可信的映射结果(最多3个点)。
进一步的说明,假设一个以数据点(x_i)为中心,半径为r的m维球(三维空间就是球),其体积是按(r^m)增长的,假设数据点是在m维球中均匀分布的,我们来看看其他数据点与(x_i)的距离随维度增大而产生的变化。
从上图可以看到,随着维度的增大,大部分数据点都聚集在m维球的表面附近,与点(x_i)的距离分布极不均衡。如果直接将这种距离关系保留到低维,就会出现拥挤问题。
怎么解决crowding问题呢?
Cook et al.(2007) 提出一种slight repulsion的方式,在基线概率分布(uniform background)中引入一个较小的混合因子(\rho),这样(q_{ij})就永远不会小于(\frac{2 \rho}{n(n-1)}) (因为一共了n(n-1)个pairs),这样在高维空间中比较远的两个点之间的(q_{ij})总是会比(p_{ij})大一点。这种称之为UNI-SNE,效果通常比标准的SNE要好。优化UNI-SNE的方法是先让(\rho)为0,使用标准的SNE优化,之后用模拟退火的方法的时候,再慢慢增加(\rho).
直接优化UNI-SNE是不行的(即一开始(\rho)不为0),因为距离较远的两个点基本是一样的(q_{ij})(等于基线分布), 即使(p_{ij})很大,一些距离变化很难在(q_{ij})中产生作用。也就是说优化中刚开始距离较远的两个聚类点,后续就无法再把他们拉近了。
2.3 t-SNE
对称SNE实际上在高维度下
另外一种减轻”拥挤问题”的方法:在高维空间下,在高维空间下我们使用高斯分布将距离转换为概率分布,在低维空间下,我们使用更加偏重长尾分布的方式来将距离转换为概率分布,使得高维度下中低等的距离在映射后能够有一个较大的距离。
我们对比一下高斯分布和t分布(如上图,code见probability/distribution.md), t分布受异常值影响更小,拟合结果更为合理,较好的捕获了数据的整体特征。
使用了t分布之后的q变化,如下:
[q_{ij} = \frac{(1 + \mid \mid y_i -y_j \mid \mid 2){-1}}{\sum_{k \neq l} (1 + \mid \mid y_i -y_j \mid \mid 2){-1}}]
此外,t分布是无限多个高斯分布的叠加,计算上不是指数的,会方便很多。优化的梯度如下:
[\frac{\delta C}{\delta y_i} = 4 \sum_j(p_{ij}-q_{ij})(y_i-y_j)(1+ \mid \mid y_i-y_j \mid \mid 2){-1}]
t-sne的有效性,也可以从上图中看到:横轴表示距离,纵轴表示相似度, 可以看到,对于较大相似度的点,t分布在低维空间中的距离需要稍小一点;而对于低相似度的点,t分布在低维空间中的距离需要更远。这恰好满足了我们的需求,即同一簇内的点(距离较近)聚合的更紧密,不同簇之间的点(距离较远)更加疏远。
总结一下,t-SNE的梯度更新有两大优势:
- 对于不相似的点,用一个较小的距离会产生较大的梯度来让这些点排斥开来。
- 这种排斥又不会无限大(梯度中分母),避免不相似的点距离太远。
2.4 算法过程
算法详细过程如下:
- Data: (X = {x_1, … , x_n})
- 计算cost function的参数:困惑度Perp
- 优化参数: 设置迭代次数T, 学习速率(\eta), 动量(\alpha(t))
- 目标结果是低维数据表示 (Y^T = {y_1, … , y_n})
- 计算在给定Perp下的条件概率(p_{j \mid i})(参见上面公式)
- 令 (p_{ij} = \frac{p_{j \mid i} + p_{i \mid j}}{2n})
- 用 (N(0, 10^{-4}I)) 随机初始化 Y
- 迭代,从 t = 1 到 T, 做如下操作:
- 计算低维度下的 (q_{ij})(参见上面的公式)
- 计算梯度(参见上面的公式)
- 更新 (Y^{t} = Y^{t-1} + \eta \frac{dC}{dY} + \alpha(t)(Y^{t-1} - Y^{t-2}))
优化过程中可以尝试的两个trick:
- 提前压缩(early compression):开始初始化的时候,各个点要离得近一点。这样小的距离,方便各个聚类中心的移动。可以通过引入L2正则项(距离的平方和)来实现。
- 提前夸大(early exaggeration):在开始优化阶段,(p_{ij})乘以一个大于1的数进行扩大,来避免因为(q_{ij})太小导致优化太慢的问题。比如前50次迭代,(p_{ij})乘以4
优化的过程动态图如下:
2.5 不足
主要不足有四个:
- 主要用于可视化,很难用于其他目的。比如测试集合降维,因为他没有显式的预估部分,不能在测试集合直接降维;比如降维到10维,因为t分布偏重长尾,1个自由度的t分布很难保存好局部特征,可能需要设置成更高的自由度。
- t-SNE倾向于保存局部特征,对于本征维数(intrinsic dimensionality)本身就很高的数据集,是不可能完整的映射到2-3维的空间
- t-SNE没有唯一最优解,且没有预估部分。如果想要做预估,可以考虑降维之后,再构建一个回归方程之类的模型去做。但是要注意,t-sne中距离本身是没有意义,都是概率分布问题。
- 训练太慢。有很多基于树的算法在t-sne上做一些改进
后续有机会补充。
- multiple maps of t-SNE
- parametric t-SNE
- Visualizing Large-scale and High-dimensional Data
4.参考文档
- Maaten, L., & Hinton, G. (2008). Visualizing data using t-SNE. Journal of Machine Learning Research.
文中的插图绘制:
1. # coding:utf-8
2.
3. import numpy as np
4. from numpy.linalg import norm
5. from matplotlib import pyplot as plt
6. plt.style.use(‘ggplot’)
7.
8. def sne_crowding():
9. npoints = 1000 # 抽取1000个m维球内均匀分布的点
10. plt.figure(figsize=(20, 5))
11. for i, m in enumerate((2, 3, 5, 8)):
12. # 这里模拟m维球中的均匀分布用到了拒绝采样,
13. # 即先生成m维立方中的均匀分布,再剔除m维球外部的点
14. accepts = []
15. while len(accepts) < 1000:
16. points = np.random.rand(500, m)
17. accepts.extend([d for d in norm(points, axis=1)
18. if d <= 1.0]) # 拒绝采样
19. accepts = accepts[:npoints]
20. ax = plt.subplot(1, 4, i+1)
21. if i 0:
22. ax.set_ylabel(‘count’)
23. if i 2:
24.
25. ax.set_xlabel(‘distance’)
26. ax.hist(accepts, bins=np.linspace(0., 1., 50))
27. ax.set_title(‘m=%s’ %m)
28. plt.savefig(“./images/sne_crowding.png”)
29.
30. x = np.linspace(0, 4, 100)
31. ta = 1 / (1 + np.square(x))
32. tb = np.sum(ta) - 1
33. qa = np.exp(-np.square(x))
34. qb = np.sum(qa) - 1
35.
36. def sne_norm_t_dist_cost():
37. plt.figure(figsize=(8, 5))
38. plt.plot(qa/qb, c=“b”, label=“normal-dist”)
39. plt.plot(ta/tb, c=“g”, label=“t-dist”)
40. plt.plot((0, 20), (0.025, 0.025), ‘r–’)
41. plt.text(10, 0.022, r‘’)42. plt.text(20, 0.026, r‘’)43.
44. plt.plot((0, 55), (0.005, 0.005), ‘r–’)
45. plt.text(36, 0.003, r‘’)46. plt.text(55, 0.007, r‘’)47.
48. plt.title(“probability of distance”)
49. plt.xlabel(“distance”)
50. plt.ylabel(“probability”)
51. plt.legend()
52. plt.savefig(“./images/sne_norm_t_dist_cost.png”)
53.
54. if name ‘main’:
55. sne_crowding()
56. sne_norm_t_dist_cost()附录一下t-sne的完整代码实现:
1. # coding utf-8
2. ‘’‘
3. 代码参考了作者Laurens van der Maaten的开放出的t-sne代码, 并没有用类进行实现,主要是优化了计算的实现
4. ’‘’
5. import numpy as np
6.
7.
8. def cal_pairwise_dist(x):
9. ‘’‘计算pairwise 距离, x是matrix
10. (a-b)^2 = a^w + b^2 - 2ab
11. ‘’’
12. sum_x = np.sum(np.square(x), 1)
13. dist = np.add(np.add(-2 np.dot(x, x.T), sum_x).T, sum_x)
14. return dist
15.
16.
17. def cal_perplexity(dist, idx=0, beta=1.0):
18. ‘’‘计算perplexity, D是距离向量,
19. idx指dist中自己与自己距离的位置,beta是高斯分布参数
20. 这里的perp仅计算了熵,方便计算
21. ‘’’
22. prob = np.exp(-dist beta)
23. # 设置自身prob为0
24. prob[idx] = 0
25. sum_prob = np.sum(prob)
26. perp = np.log(sum_prob) + beta np.sum(dist prob) / sum_prob
27. prob /= sum_prob
28. return perp, prob
29.
30.
31. def seach_prob(x, tol=1e-5, perplexity=30.0):
32. ‘’‘二分搜索寻找beta,并计算pairwise的prob
33. ‘’’
34.
35. # 初始化参数
36. print(“Computing pairwise distances…”)
37. (n, d) = x.shape
38. dist = cal_pairwise_dist(x)
39. pair_prob = np.zeros((n, n))
40. beta = np.ones((n, 1))
41. # 取log,方便后续计算
42. base_perp = np.log(perplexity)
43.
44. for i in range(n):
45. if i % 500 0:
46. print(“Computing pair_prob for point %s of %s …” %(i,n))
47.
48. betamin = -np.inf
49. betamax = np.inf
50. perp, this_prob = cal_perplexity(dist[i], i, beta[i])
51.
52. # 二分搜索,寻找最佳sigma下的prob
53. perp_diff = perp - base_perp
54. tries = 0
55. while np.abs(perp_diff) > tol and tries < 50:
56. if perp_diff > 0:
57. betamin = beta[i].copy()
58. if betamax np.inf or betamax -np.inf:
59. beta[i] = beta[i] 2
60. else:
61. beta[i] = (beta[i] + betamax) / 2
62. else:
63. betamax = beta[i].copy()
64. if betamin np.inf or betamin -np.inf:
65. beta[i] = beta[i] / 2
66. else:
67. beta[i] = (beta[i] + betamin) / 2
68.
69. # 更新perb,prob值
70. perp, this_prob = cal_perplexity(dist[i], i, beta[i])
71. perp_diff = perp - base_perp
72. tries = tries + 1
73. # 记录prob值
74. pair_prob[i,] = this_prob
75. print(“Mean value of sigma: “, np.mean(np.sqrt(1 / beta)))
76. return pair_prob
77.
78.
79. def pca(x, no_dims = 50):
80. ‘’’ PCA算法
81. 使用PCA先进行预降维
82. ‘’'
83. print(“Preprocessing the data using PCA…”)
84. (n, d) = x.shape
85. x = x - np.tile(np.mean(x, 0), (n, 1))
86. l, M = np.linalg.eig(np.dot(x.T, x))
87. y = np.dot(x, M[:,0:no_dims])
88. return y
89.
90.
91. def tsne(x, no_dims=2, initial_dims=50, perplexity=30.0, max_iter=1000):
92. ””“Runs t-SNE on the dataset in the NxD array x
93. to reduce its dimensionality to no_dims dimensions.
94. The syntaxis of the function is Y = tsne.tsne(x, no_dims, perplexity),
95. where x is an NxD NumPy array.
96. “””
97.
98. # Check inputs
99. if isinstance(no_dims, float):
100. print(“Error: array x should have type float.”)
101. return -1
102. if round(no_dims) != no_dims:
103. print(“Error: number of dimensions should be an integer.”)
104. return -1
105.
106. # 初始化参数和变量
107. x = pca(x, initial_dims).real
108. (n, d) = x.shape
109. initial_momentum = 0.5
110. final_momentum = 0.8
111. eta = 500
112. min_gain = 0.01
113. y = np.random.randn(n, no_dims)
114. dy = np.zeros((n, no_dims))
115. iy = np.zeros((n, no_dims))
116. gains = np.ones((n, no_dims))
117.
118. # 对称化
119. P = seach_prob(x, 1e-5, perplexity)
120. P = P + np.transpose(P)
121. P = P / np.sum(P)
122. # early exaggeration
123. P = P 4
124. P = np.maximum(P, 1e-12)
125.
126. # Run iterations
127. for iter in range(max_iter):
128. # Compute pairwise affinities
129. sum_y = np.sum(np.square(y), 1)
130. num = 1 / (1 + np.add(np.add(-2 np.dot(y, y.T), sum_y).T, sum_y))
131. num[range(n), range(n)] = 0
132. Q = num / np.sum(num)
133. Q = np.maximum(Q, 1e-12)
134.
135. # Compute gradient
136. PQ = P - Q
137. for i in range(n):
138. dy[i,:] = np.sum(np.tile(PQ[:,i] num[:,i], (no_dims, 1)).T (y[i,:] - y), 0)
139.
140. # Perform the update
141. if iter < 20:
142. momentum = initial_momentum
143. else:
144. momentum = final_momentum
145. gains = (gains + 0.2) ((dy > 0) != (iy > 0)) + (gains 0.8) ((dy > 0) (iy > 0))
146. gains[gains < min_gain] = min_gain
147. iy = momentum iy - eta (gains * dy)
148. y = y + iy
149. y = y - np.tile(np.mean(y, 0), (n, 1))
150. # Compute current value of cost function
151. if (iter + 1) % 100 0:
152. if iter > 100:
153. C = np.sum(P np.log(P / Q))
154. else:
155. C = np.sum( P/4 np.log( P/4 / Q))
156. print("Iteration “, (iter + 1), ”: error is ", C)
157. # Stop lying about P-values
158. if iter 100:
159. P = P / 4
160. print(“finished training!”)
161. return y
162.
163.
164. if name “main”:
165. # Run Y = tsne.tsne(X, no_dims, perplexity) to perform t-SNE on your dataset.
166. X = np.loadtxt(“mnist2500_X.txt”)
167. labels = np.loadtxt(“mnist2500_labels.txt”)
168. Y = tsne(X, 2, 50, 20.0)
169. from matplotlib import pyplot as plt
170. plt.scatter(Y[:,0], Y[:,1], 20, labels)
171. plt.show()Recommend
About Joyk
Aggregate valuable and interesting links.
Joyk means Joy of geeK