

OBIA:900+ 患者、193w+ 影像,中科院基因组所发布我国首个生物影像共享数据库
source link: https://blog.csdn.net/HyperAI/article/details/133748404
Go to the source link to view the article. You can view the picture content, updated content and better typesetting reading experience. If the link is broken, please click the button below to view the snapshot at that time.

OBIA:900+ 患者、193w+ 影像,中科院基因组所发布我国首个生物影像共享数据库

看病就医,拍片已是常例。CT、核磁、X 光等影像资料可以用非侵入式手段透过人体,使内部器官、组织状况清晰可见,为临床诊断和疾病治疗提供可靠依据。
随着医学影像技术广泛发展,影像资料已占据国内医疗数据的 80% 以上,影像科医生供不应求、各级医院诊断结果存在差异、医疗资源分配不均等痛点也日益凸显。
AI 结合医学影像具有非常大的想象空间,感觉认知及深度学习技术在识别医疗影像诊断结果方面拥有人类无可比拟的优势,可辅助医生降低误诊率、提高工作效率。
然而,高质量的 AI 算法需要足够大且有代表性的图像数据集, 这些医疗图像又往往涉及大量敏感隐私信息,加上各级医院之间存在「数据孤岛」,不完整的共享系统使得医疗影像 AI 的可用资源有限。
作者 | 铁塔
编辑 | 三羊、雪菜
全球已有不少国家建设了各类医学影像数据共享数据库,我国在此领域同国际社会仍有差距,为推动高质量的医学生物影像数据共享,中科院基因组所(中国国家生物信息中心)建立了开放生物医学成像档案 (OBIA)。
作为国内首个开放的生物医学成像数据和相关临床数据存储库, OBIA 对全球医疗从业者及相关学者免费开放。相关成果预印版已于 2023 年 9 月 25 日发表在 「bioRxiv」。

**论文链接: **https://www.nature.com/articles/s42256-023-00704-7
关注「HyperAI超神经」公众号,后台回复「OBIA」获取论文完整 PDF
OBIA 数据库建设及实施过程
作为中国国家生物信息中心的核心数据库资源,OBIA 接受来自世界各地的图像提交并提供所有公开数据的免费开放访问,它支持对影像数据的去标识化 (de-identification)、管理和质量控制 (quality control), 提供浏览、检索和下载等数据服务,可促进现有图像数据和临床数据的重复利用。
OBIA 采用 5 种数据对象 (Collection, Individual, Study, Series, Image) 进行数据组织,接受多模态、多器官、多疾病的生物医学图像提交。
为保护个人隐私,OBIA 制定了统一的去标识化和质量控制流程, 并为数据提交、浏览和检索以及图像检索,提供直观友好的 Web 界面。总体来讲,OBIA 为国内生物医学成像数据管理提供了一个可靠的平台,有助于支持全球生物医学研究。
图 1:OBIA 访问界面
访问地址:https://ngdc.cncb.ac.cn/obia
实现细节——图像检索
深度神经网络擅长提取优势特征, 可用于检索人体各器官的多模态医学图像,并在小样本情况下提高排序性能。与传统方法相比,基于深度学习的方法如尺度不变特征转换 (SIFT)、局部二值模式 (LBP) 和定向梯度直方图 (HOG) 能够表现出更好的性能。
在 OBIA,研究人员以癌症影像数据库 TCIA 的多模态癌症数据为基础,将 EfficientNet 用作特征提取器,使用三元组网络和注意力模块 (attention module) 来训练模型,并将图像压缩为离散哈希值 (图 2)。随后,为加快推理性能并减少推理延迟,训练好的模型被转换为 TensorRT 格式,使用 Faiss 存储哈希码。
研究人员利用汉明距离 (Hamming distance) 计算图像相似度,并返回最相似的图像,结果表明,该模型的平均精度 (MAP) 值超过了 TCIA 数据集上现有高级图像检索模型的性能。
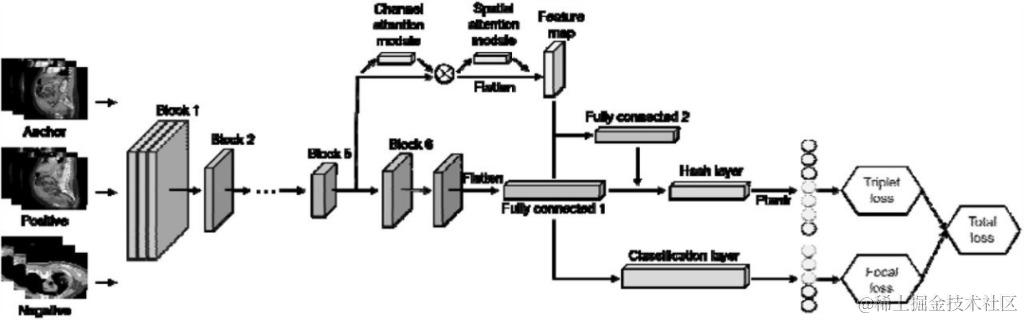
图 2:基于注意力和层融合模块的深度三元组哈希
该模型以 EfficientNet-B6 为主网络,利用 Block5 中的 CBAM 注意模块获取特征映射。在全连接层中采用层融合,利用焦点损失和三元组损失生成哈希码和类嵌入。
● CBAM:convolutional block attention module,卷积块注意力模块
● EfficientNet:Google 于 2019 年提出的新型 CNN 网络,具备极高的参数效率和速度,在图片分类领域表现优异
● Faiss:Facebook 人工智能研究院开发的高性能相似性搜索库,通常用于深度学习
数据库内容及使用——数据模型
如图 3 所示,OBIA 中的成像数据分为 5 种对象类型: Collection, Individual, Study, Series, Image,分别指:
• Collections: 以「OBIA」为前缀,提供完整提交的总体描述;
• Individual: 登记编号以「 I 」为前缀,定义接受或登记接受医疗保健服务的人类或非人类生物体特征;
• Study: 采用以「S」为前缀的登录号,包含对个人进行放射检查的描述性信息;
• Series: 研究可以根据不同的逻辑(如身体部位或方向)分成一个或多个 Series;
• Image: 描述单个 DICOM 文件(Digital Imaging and Communications in Medicine,医学数字成像和通信)的像素数据,Image 与单个 Study 中的单个 Series 相关。
注:DICOM 是一种广泛应用于医学影像领域的国际标准,定义了一套存储、传输、共享和打印医学影像数据的规范和协议,使不同厂商生产的医学设备和软件之间可以相互兼容和交流。
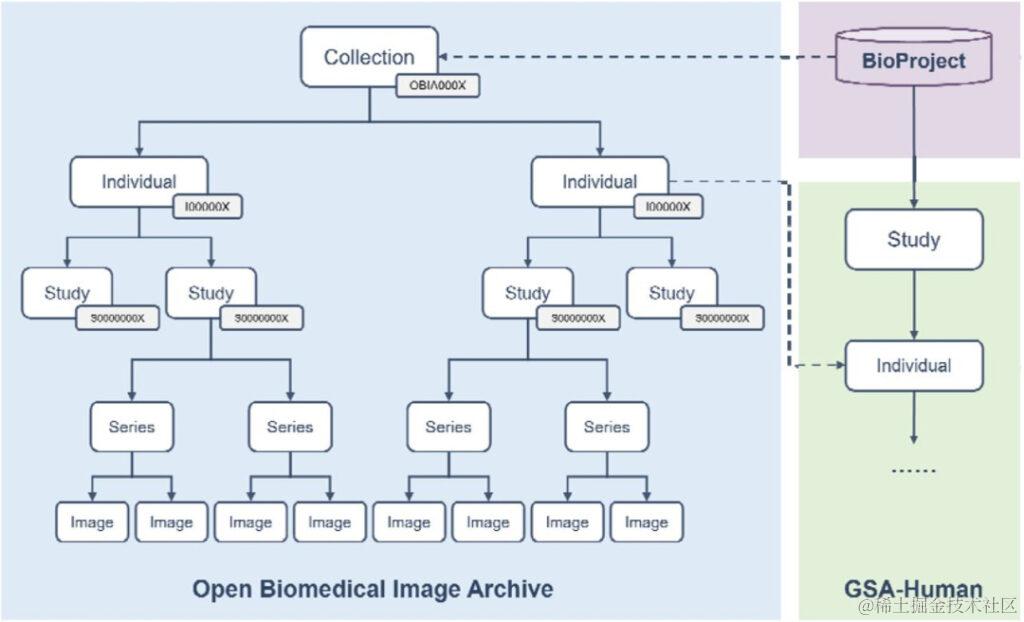
图 3:OBIA 数据模型
基于这些标准化的数据对象,OBIA 将 DICOM 标准定义的图像结构与实际研究项目连接起来, 实现了数据的共享和交换。
此外,OBIA中的每个 Collections 都链接到 BioProject 以提供有关研究项目的描述性元数据;
如若可行,OBIA 的 Individual 可通过 Individual 登录号与 GSA-Human 相关联,后者将成像数据与基因组数据联系起来,供研究人员进行多组学分析。
BioProject 链接地址:
https://ngdc.cncb.ac.cn/bioproject/
GSA-Human 链接地址:
https://ngdc.cncb.ac.cn/gsa-human/
数据库内容及使用——去标识化和质量控制
生物医疗图像可能包含受保护的健康信息 (PHI,Protected Health Information),需要经过适当处理以尽量降低侵犯个人隐私的风险。为了在删除 PHI 的同时尽可能多地保留有价值的科学信息,OBIA 提供了一个符合 DICOM 标准的去标识化和质量控制机制 (图 4)。
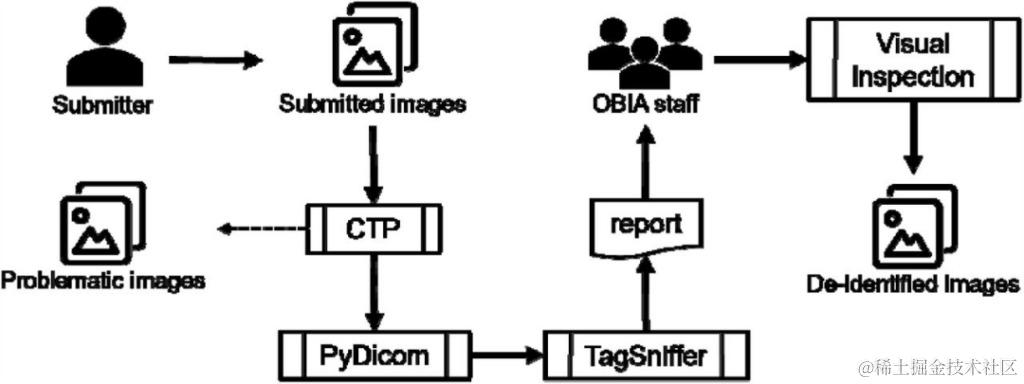
图 4:OBIA 去标识化和质量控制机制
OBIA 利用北美放射学会 (RSNA) 的 MIRC 临床试验处理器 (CTP) 进行大部分去标识化工作:
• 对于标准标记 (standared tags), 研究人员构建了一个 CTP ,并开发了一个通用的基础去标识化脚本,用于删除或隐去某些包含或可能包含 PHI 的标准标记;
• 对于私有标记 (private tags), 使用 PyDicom 进行处理,保留其纯数字属性。
去标识过程结束后,OBIA 开始运行质量控制程序:
• 有问题的图像: 隔离图像,提交者可以提供相关信息对图像进行修复或完全抛弃(该类图像是指带有空白标题或缺少患者 ID、损坏、混合了其他患者图像等类型的图像);
• 重复的图像: 只保留一个。
随后 OBIA 使用 TagSniffer 为所有图像生成一个报告,报告中所有 DICOM 元素都经过仔细审查,以确保它们不包含 PHI,并且某些值(例如患者 ID、研究日期)按照预期进行修改。
此外,OBIA 工作人员还会对图像像素执行目视检查, 以确保像素值中没有包含 PHI,并且图像是可见和未损坏的。
数据库内容及使用——数据统计
截至 2023 年 9 月,OBIA 共收集了 937 个「Individual」、4,136 个「Study」、24,701 个「Series」和 1,938,309 张「Image」,涵盖 9 种模态和 30 个解剖部位。
具有代表性的成像模态包括 X 射线计算机断层成像 (CT)、磁共振 (MR) 和数字 X 线摄影 (DX),解剖部位包括腹部、胸部、胸部、头部、肝脏、骨盆等。
第 1 批提交给 OBIA 的资料来自 301 医院, 包括 3 种主要妇科肿瘤(子宫内膜癌、卵巢癌和宫颈癌)的影像数据。
如表 1 所示,这些数据被划进 4 个「Collections」,列示了「Individual」数量、「Study」数量、「Series」数量和「Image」数量。此外,OBIA 还收集了相关临床元数据, 如人口统计学数据、病史、家族史、诊断、病理类型和治疗方法等。
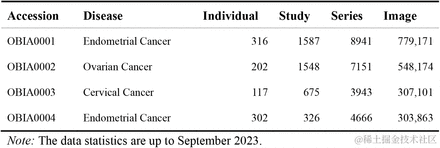
表 1:第 1 批提交给 OBIA 的资料
打破数据藩篱,国内外构建医疗数据共享平台
数据只有在流通中才会产生价值,为提升生物影像数据共享水平,全球不少国家致力于开放医疗数据库的建设:
• 美国国立卫生研究院 (NIH): 赞助了若干知识库,如新冠肺炎相关医学影像和数据的开放获取平台 MIDRC,收集神经和脑成像的 IDA, NITRC-IR, FITBIR, OpenNeuro 和 NDA,癌症影像数据库 TCIA 和 IDC(其中 TCIA 在本地提供图像,IDC 在癌症研究数据共享云环境中提供图像);
• 英国癌症研究中心 (cancer Research UK): 赞助了 OPTIMAM 乳房 X 线摄影图像数据库 (OMI-DB);
• 葡萄牙波尔图大学 (University of Porto): 赞助了乳腺癌数字存储库 (BCDR),提供带注释的乳腺癌图像和临床细节;
以上存储库中,除了 NITRC-IR 和 IDC,其他大多数都支持数据去标识化和质量控制。 此外,一些大学或机构也提供开源数据集,如 OASIS, EchoNet-Dynamic, CAMUS project 等。
图 5:MIDRC 数据库内一位 79 岁患者的胸部 CT
在国内,华中科技大学提供了 COVID-19 的整合 CT 图像和 CFs 的开放资源, 包括肺炎(含新冠肺炎)患者的 CT 图像和临床特征,但仅限于单一疾病,可用研究资源有限,因此目前国内仍然缺乏专门存储和接受各种疾病及模态资料提交的数据库。
中科院基因组建立的 OBIA 填补了国内生物医疗影像数据开放共享的空白,方便不同机构的研究人员共享临床相关成像数据,可有效弥合中国在生物医学成像数据库领域的差距。
研究人员在论文中表示,未来将持续升级 OBIA 的基础设施,加大安全防护措施,同时将收集更多类型生物医学影像数据,扩大数据源,多措并举不断向「保留尽可能多的有效图像元数据,为科研人员提供高质量的成像数据」的目标迈进。
—— 完 ——
Recommend
About Joyk
Aggregate valuable and interesting links.
Joyk means Joy of geeK